
REDEFAR es una estructura coordinada en las diferentes etapas del descubrimiento temprano de fármacos, añadiendo valor en cada una de ellas y reduciendo el riesgo intrínseco a este tipo de actividades que implican ciencia disruptiva con una aproximación al mercado innovadora y acorde con los requerimientos de la industria farmacéutica y biotecnológica, con el objetivo de agilizar el proceso de descubrimiento de nuevos fármacos.
Áreas Transversales
Formación
Información
Management
Difusión y explotación de resultados
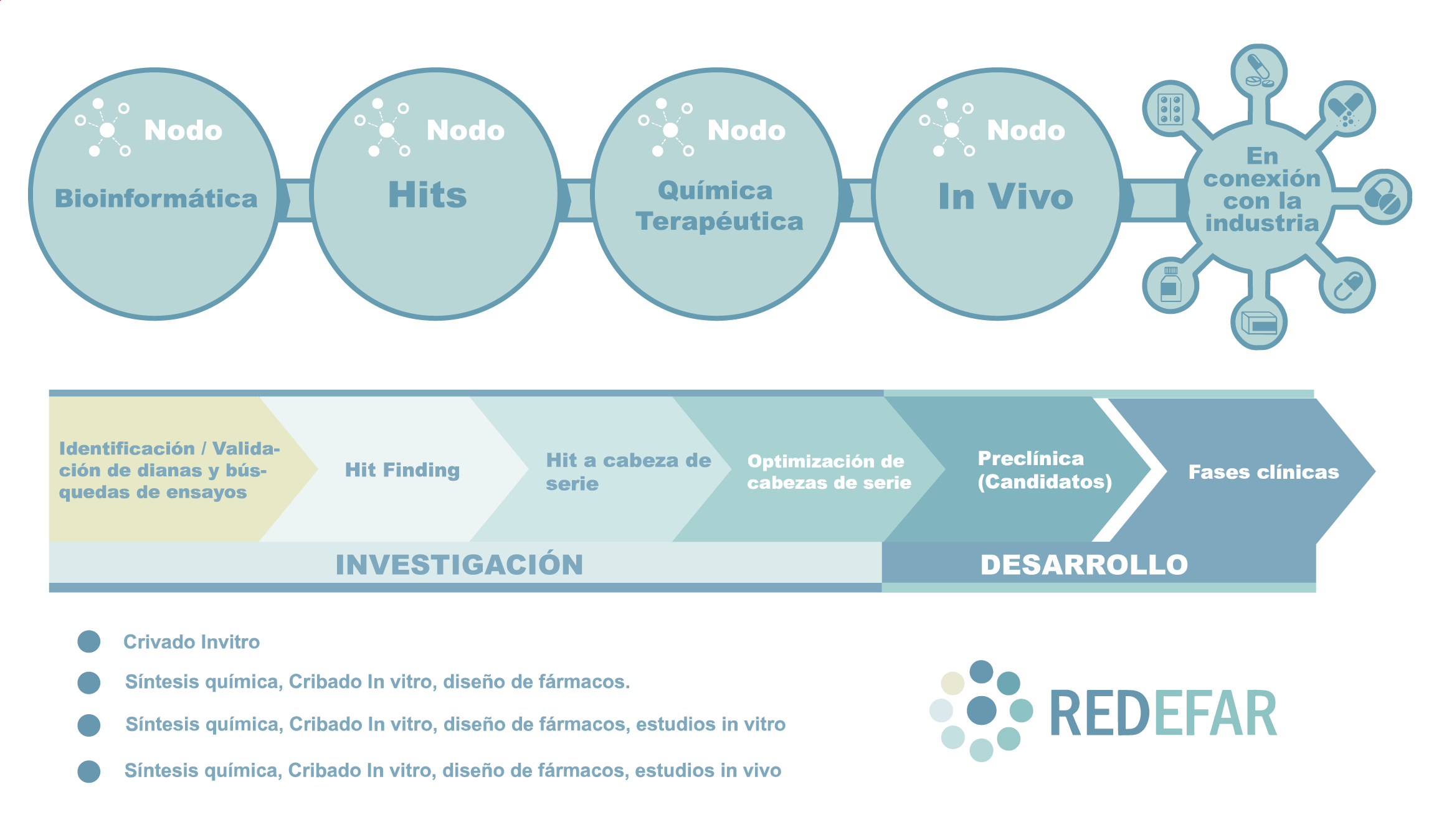
Nodos
La Fundación MEDINA es un Centro de Investigación sin ánimo de lucro para el descubrimiento de nuevos fármacos que respondan a necesidades médicas no cubiertas. Establecido en el Parque Tecnológico de Ciencias de la Salud (PTS) de Granada en 2008 a partir de la alianza público-privada de la Junta de Andalucía, La Universidad de Granada y MSD de España, MEDINA surge a partir del proyecto desarrollado en el antiguo centro de investigación básica de MSD España (CIBE) con más de 50 años de experiencia en el descubrimiento de nuevos fármacos a partir de productos naturales y la evaluación preclínica de nuevos candidatos.
El grupo de la Fundación MEDINA incluye investigadores con una larga experiencia en el sector farmacéutico, la industria biotech y centros académicos, y opera como un equipo multidisciplinar con capacidades en los campos de la biología celular y molecular, la microbiología, la química de productos naturales, la gestión de compuestos, automatización y bioinformática.
MEDINA posee una de las mayores librerías de extractos productos naturales de origen microbiano (130.000 muestras) para el screening de nuevos fármacos en su plataforma de cribado de alta densidad, MEDINA mantiene un amplio número de colaboraciones con grupos de investigación nacionales e internacionales, parte de las cuales se integran dentro de consorcios europeos FP7 e IMI en los que participa así como proyectos con empresas farmacéuticas y biotecnológicas. MEDINA es miembro asociado de la Alianza BEAM de empresas biotecnológicas para el descubrimiento de nuevos antibióticos, y miembro fundador de la nueva Alianza Española para el Descubrimiento de nuevos Antibióticos AD-ES.
Es una de las siete plataformas de screening de altas capacidades de la Red Europea ERIC EU-OPENSCREEN.
El grupo de investigación BioFarma/Plataforma Innopharma de la USC, se dedica desde 1998 a la investigación académica aplicada a descubrimiento temprano de fármacos, en colaboración con la industria farmacéutica y biotecnológica. Ha desarrollado 25 programas en el ámbito de descubrimiento de fármacos, los cuales están incluidos en más de 90 proyectos y contratos financiados por organismos públicos y privados, de los cuales 15 compuestos han llegado a ensayos clínicos en humanos. El grupo pone a disposición de la investigación pública en España una quimioteca de más de 60000 compuestos incluyendo fármacos aprobados en Europa, EEUU y Japón para reposicionamiento, compuestos exclusivos, subquimiotecas focalizadas en distintas familias de dianas y compuestos seleccionados a partir de catálogos de proveedores comerciales en base a propiedades lead-like cubriendo el máximo espacio estructural y biológico a partir de estudios ″in silico″. La plataforma se ha especializado en:
- Farmacogenómica, con laboratorios para la ejecución de campañas de cribado de alto rendimiento (High Throughput Screening-HTS y High Content Screening-HCS) junto con laboratorios de secuenciación, ultrasecuenciación y expresión de ácidos nucleicos, para la búsqueda de dianas y de biomarcadores de respuesta terapéutica, siendo referencia como Beta tester de nuevos ensayos para empresas desarrolladoras;
- Acciones de catalizador de innovación académica y traslado a valor en descubrimiento de fármacos, gracias a la calidad científico-técnica del equipo humano interdisciplinar, formado en los mejores centros internacionales de investigación en descubrimiento de fármacos.
Es una de las siete plataformas de screening de altas capacidades de la Red Europea ERIC EU-OPENSCREEN.
La plataforma de Cribado Farmacológico del CIPF constituye un servicio tecnológico destinado a la evaluación biológica/farmacológica de librerías de compuestos propias, así como procedentes de organismos externos, con el fin de obtener y caracterizar nuevos agentes bioactivos simples o en combinación bajo unas condiciones clínicas específicas.
Sus objetivos van principalmente orientados a identificar compuestos moduladores de interacciones proteína-proteína y/o proteína-RNA claves en patologías humanas, y a desarrollar ensayos en el área de las células madre adultas y embrionarias (proliferación y diferenciación). Este tipo de ensayo puede permitir el desarrollo de terapias personalizadas mediante estratificación de pacientes, así como dar soporte al desarrollo de fármacos en ensayos clínicos acelerando el proceso de translación del laboratorio al paciente.
Dispone de dos librerías de compuestos químicos (Myriad y Prestwick) de más de 11000 compuestos. También se dispone de una quimioteca de fragmentos para llevar a cabo cribados farmacológicos de forma modular mediante interacciones con proteínas seleccionadas por RMN. Esta aproximación permite la identificación de biomarcadores de diferente naturaleza. Dispone también del Laboratorio de Polímeros Terapéuticos, el primero de su clase en España, dirigido por la Dra. Vicent, y consolidado con la concesión de una ayuda ERC Consolidator Grant (MyNano).
Es una de las plataformas especializadas de la Red Europea de screening ERIC EU-OPENSCREEN.
El Laboratorio de Medicina Computacional (LMC) de la Facultad de Medicina de la Universidad Autónoma de Barcelona está trabajando en una amplia gama de proyectos centrados en la bioinformática translacional. Herramientas computacionales, que van desde métodos estadísticos y minería-de-datos, hasta técnicas de dinámica molecular, se utilizan en el análisis de genes, proteínas y sus interacciones con fármacos.
LMC posee los equipos informáticos necesarios para llevar a cabo este proyecto. El laboratorio cuenta actualmente con una red informática de uso personal que contiene catorce servidores con 48-core AMD Interlagos (672 núcleos en total) y quince servidores con 20-core Intel Xeon E5 V3 y dos tarjetas gráficas NVIDIA GTX980 (300 núcleos + 30 GTX980 en total) para el cálculo intensivo. Además, tiene acceso al superordenador Marenostrum en el Barcelona Supercomputing Center. La gran cantidad de datos generados en este tipo de estudios se almacena en un servidor de datos NetApp (48 Tb). Asimismo, LMC tiene instalado software de bioinformática: MOE, Chemical Computing Group; Discovery Studio, Accelrys; PyMOL, Schrödinger; LigandScout, Inteligand; GRASP, UCLA; GOLD, Cambridge Crystallographic Data Centre; AMBER, UCSF; NAMD, University of Illinois; GROMACS; GAUSSIAN, Gaussian Inc; SPSS, SPSS Inc.
Instituto Hospital del Mar de Investigaciones Médicas
Ferrán Sanz
Gestión del conocimiento y predicción toxicológica
El Programa de Investigación en Informática Biomédica (GRIB) del Instituto Hospital del Mar de Investigaciones Médicas (IMIM) y de la Universidad Pompeu Fabra (UPF). El GRIB lleva a cabo investigación fundamental y desarrollo de herramientas sobre la aplicación de las tecnologías de la información y de los métodos computacionales en las ciencias de la salud y de la vida. El GRIB está compuesto por más de 60 científicos y por personal técnico y de gestión. Entre los grupos de investigación del GRIB están los de Farmacoinformática (M. Pastor), Farmacología de Sistemas (J. Mestres) e Informática Biomédica Integrada (L. Furlong) que participarán especialmente en la presente red.
El GRIB tiene una dilatada experiencia de colaboración con la industria farmacéutica y biotecnológica. El GRIB es el nodo de Informática Biomédica del Instituto Nacional de Bioinformática (INB) y es socio de la la plataforma europea de bioinformática ELIXIR-EXCELLERATE.
Su coordinador es coordinador académico del proyecto eTOX sobre la predicción in silico de la toxicidad de medicamentos (http://www.e-tox.net/) . Además, es o ha sido socio en muchos otros proyectos financiados por la Unión Europea (ver CV) y es el coordinador académico de la Plataforma Española de Medicamentos Innovadores (PTEMI) y Director Científico de Bioinformatics Barcelona (BiB).
El Laboratorio de Química Médica de la Universidad Complutense de Madrid(http://www.ucm.es/info/quimicamedica), dirigido por la Catedrática María Luz López Rodríguez, se compone de 17 miembros. Los investigadores que componen el grupo poseen una amplia formación y experiencia en el área de la química médica y una demostrada capacidad para abordar tanto la investigación básica como la aplicada y traslacional, habiendo obtenido valiosos avances en las áreas de la química médica y la química biológica (progresión de agonistas 5-HT1A hasta fase clínica, validación de las enzimas FASN e ICMT como dianas oncológicas, descubrimiento mediante sondas químicas de un nuevo receptor cannabinoide mitocondrial, desarrollo de un modulador alostérico del receptor GLP1 para el tratamiento de la diabetes tipo 2, validación de la enzima MAGL como diana para la esclerosis múltiple).
Como prueba del interés terapéutico de los resultados generados, los miembros del grupo son inventores de 6 patentes en explotación actual por la industria farmacéutica y en los últimos diez años hemos firmado 10 contratos de colaboración con empresas del sector UCB Pharma, Exonhit Therapeutics, Italfarmaco y ViviaBiotech en el marco del programa INNPACTO).
El Grupo de Química Médica y Biológica Traslacional, ubicado en el Centro de Investigaciones Biológicas del Consejo Superior de Investigaciones Científicas (http://www.chemphysbiol.org/research), es un grupo multidisciplinar formado por químicos, farmacéuticos y biólogos cuyo objetivo es el diseño y desarrollo de fármacos innovadores para enfermedades neurológicas y parasitarias. Cuenta con experiencia en modelado molecular y quimioinformática, síntesis orgánica, cribado biológico, optimización de propiedades ADMETox, y gestión empresarial.
Utiliza habitualmente tecnologías punteras como genética química, química combinatoria dinámica, síntesis en fase sólida, etc., y cuida nuestros procedimientos para situarnos en la ″química verde″ y sostenible, aplicando biocatalisis o síntesis asistida por microondas. El grupo cuenta con una quimioteca propia de estructuras privilegiadas para el sistema nervioso central.
Su investigación, frontera entre la ciencia básica para el descubrimiento de nuevos fármacos y la aplicada, tiene un alto contenido traslacional.
Los programas de investigación del grupo se diseñan desde las fases tempranas del descubrimiento de nuevos fármacos hasta la prueba de eficacia en modelos animales representativos. Las colaboraciones científicas así como la transferencia de tecnología hacia empresas del sector farmacéutico son clave en la consecución de los objetivos. La política interna de grupo lleva a patentar los resultados previos a su difusión científica y a trabajar activamente en la transferencia de los mismos. En Febrero de 2014 se fundó la spin-off ANKAR PHARMA, que nació para apoyar la transferencia de los resultados de investigación en nuevos fármacos desde el laboratorio hasta las fases clínicas.
El grupo de investigación en Obesidómica Funcional y Metabolismo Molecular dirigido por este investigador en la USC es uno de los líderes europeos en el campo de la fisiopatología de la obesidad. Carlos Diéguez posee más de 400 publicaciones, más de 8.000 citas y un índice-H de 65 (ISI Web of Knowledge). Destaca no sólo en publicaciones y captación de financiación, si no también en formación de investigadores punteros: ha dirigido 22 tesis y ha contribuído a la captación de dos Starting Grants del European Research Council (ERC) por investigadores formados en su laboratorio.
El grupo de investigación está vinculado con el Centro de Biología Experimental de Galicia (CEBEGA), de la USC, que se está constituyendo como uno de los centros de referencia en experimentación animal de España.
El grupo dispone de la tecnología del más alto nivel para estudios in vivo de valoración de dianas relacioandas con balance energético y homeostasis metabólica. Las más destacables son:
- Sistema de calorimetria indirecta de última generación (TSE, Alemania) que permite valorar de forma precisa ingesta de agua/comida, actividad locomotra, y gasto energético cada 30 min durante una semana. El sistema disponible permite analizar 24 animales de forma simultánea y a las condiciones de temperatura que se deseen.
- Cámara termográfica para cuantificación de temperatura del BAT.
- Sistema de valoración de homeostasis de la glucosa mediante clamps en pequeños animales (rata/ratón).
- NMR para valoración de composición corporal.
- Amplia experiencia en immunoensayos para cuantificación de distintas hormonas/adipoquinas.
- Amplia experiencia en ISH, estereotaxis, canulaciones de animales en régimen freelly-moving, etc.
La tecnología descrita ha sido empleada por el grupo con éxito en publicaciones de alto impacto tipo Nat Medicine, Cell Metabolism, Gastroenterology, Diabetes etc).
El grupo de investigación de Neuropsicofarmacología pertenece al Área 5 de Medicamentos y Vacunas del Instituto de Investigación Biomédica de Málaga (IBIMA) y está vinculado a la Unidad de Gestión Clínica de Salud Mental en el Hospital Regional Universitario de Málaga. El grupo tiene más de 20 años de experiencia en investigación independiente en el campo de la farmacología, con especial atención a la neurofarmacología de los comportamientos motivados y la farmacología de la obesidad y desórdenes metabólicos asociados. El grupo ha publicado más de 200 artículos científicos incluyendo revistas científicas de primer nivel.
El grupo ha estado implicado en 15 patentes industriales, muchas de ellas en explotación por compañías independientes. En relación con los trastornos adictivos y metabólicos, nuestra aportación principal reside en el descubrimiento del papel central y periférico del lípido oleoiletanolamida (OEA), el primer agonista endógeno PPARα, como regulador del apetito/saciedad. Además, hemos desarrollado análogos sintéticos capaces de reducir la ingesta de comida y disminuir la hiperlipidemia, y hemos caracterizado las acciones periféricas de los endocannabinoides en la regulación del metabolismo energético.
Posee experiencia en pruebas de ingesta/saciedad, autoadministración de comida, preferencia de sabores, aversión y balance metabólico (cajas metabólicas), pruebas de autoadministración intravenosa y oral de drogas de abuso, pruebas de ansiedad, depresión y miedo condicionado (Elevated Plus Maze), pruebas de aprendizaje espacial y memoria (Y Maze, Morris Water Maze, Radial Arm Maze), pruebas de discriminación de neurolépticos (Prepulse Inhibition), pruebas de interacción social y de comportamiento estereotipados y pruebas de condicionamiento de lugar preferencial (CPP).









